20.11.2018
2 minutes de lecture
L’exemple de Clostridium beijerinckii DSM 6423, une souche naturellement productrice d’isopropanol
Qu’elle devienne critique dans une ou plusieurs décennies, la raréfaction des hydrocarbures fossiles est inévitable.
Leur utilisation massive a par ailleurs un impact environnemental scientifiquement démontré et cette double prise de conscience incite à accélérer les efforts au service de la transition énergétique.
Pour un certain nombre d’applications, ceci passe par le développement d’alternatives biosourcées crédibles, au plan économique et environnemental.
Les procédés industriels de production de carburant ont ouvert la voie : la production d’éthanol est ainsi réalisée par fermentation de sucre (glucose) issus des éléments de réserve des plantes (grain, tubercules etc.), et le sera bientôt via l’utilisation des résidus agricoles et forestiers (sucres de deuxième génération ou 2G). Produit à partir de ces ressources renouvelables, le bioéthanol est aujourd’hui le seul carburant liquide, obtenu à l’aide d’un procédé à faible impact carbone, pouvant se substituer à hauteur de 5 à 15 % à l’essence sans plomb pour alimenter nos moteurs à essence.
De façon similaire, la chimie biosourcée pourrait se substituer à la pétrochimie, avec des cibles de prédilection comme le propylène et l’éthylène, molécules phares de la pétrochimie car produites chaque année à plus de 100 millions de tonnes. La synthèse par un procédé vert de ces intermédiaires chimiques peut être envisagée par de nombreuses voies, en particulier par déshydratation d’alcools.
Le département Biotechnologie d’IFPEN travaille depuis quelques années sur la possibilité de produire des alcools à partir de matières premières biosourcées vial’utilisation de microorganismes fermentaires. Contrairement aux éthanoleries où sont utilisées des levures, ce sont des bactéries appartenant au genre Clostridium qui sont employées pour produire un mélange d’isopropanol, de butanol et d’éthanol, au travers d’un procédé fermentaire dit IBE. Celui-ci se rapproche du procédé ABE (pour Acétone, Butanol, Ethanol), employant des souches du même genre bactérien et industrialisé au siècle dernier pour fournir l’acétone nécessaire à la production de cordite[1].
De récents travaux, intégrant une collaboration avec l’équipe Food Biobased Research de l’Université de Wageningen (Pays-Bas), ont été consacrés à l’étude de souches capables de réaliser des fermentations de type IBE, et plus particulièrement la souche Clostridium beijerinckii DSM 6423. Très peu d’informations étant disponibles pour cette souche, il était important de constituer une base de données solide sur laquelle s’appuyer pour tenter d’améliorer des performances par ailleurs trop faibles pour un procédé industriel. Deux thèses IFPEN, dont une encore en cours, se sont donc focalisées sur l’étude du contenu génétique et des principales voies métaboliques présentes chez ce producteur naturel d’isopropanol.
Une première partie de ce travail a été récemment publiée (Mate de Gerando et al. 2016), décrivant la totalité du matériel génétique (ou génome) de la souche Clostridium beijerinckiiDSM 6423. Cette reconstruction (Figure 1) a permis de détecter, en plus du chromosome contenant l’ensemble des gènes responsables de la production d’alcools, la présence d‘éléments génétiques supplémentaires, nommés Eléments Génétiques Mobiles (ou EGM) car capables d’être transférés entre cellules.
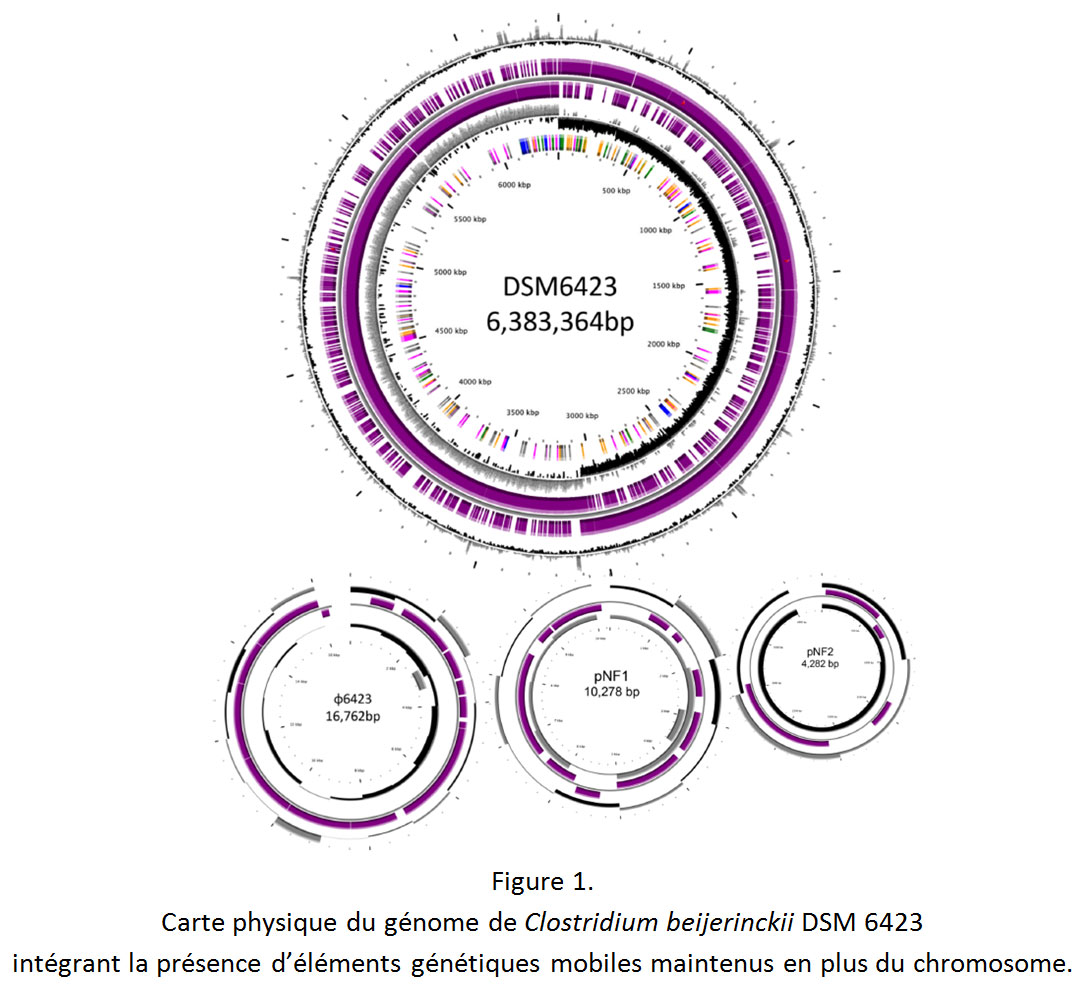
Parmi ces EGM, nous avons identifié deux plasmides[2] naturels. Ces éléments sont communs chez les bactéries, où ils participent au transfert de gènes d’intérêt entre les souches, voire entre les espèces. En revanche, la présence d’un bactériophage, maintenu sous la forme d’un ADN[3] double brin linéaire, est plus surprenante puisqu’il s’agit d’un type d’EGM qui n’avait pas encore été décrit chez une souche fermentaire de Clostridium. Le rôle de tous ces EGM est encore inconnu et doit être mieux compris car leurs présence pourrait ajouter un niveau de difficulté dans l’optique de modifier génétiquement le chromosome de cette souche.
Après avoir reconstitué la carte physique du génome, des travaux ont été menés pour analyser les mécanismes de régulation de l’expression des gènes au cours du processus de fermentation du glucose. Nous avons, pour ce faire, employé des méthodes récentes d’analyses transcriptomiques[4] capables de quantifier l’ensemble des ARN[5] d’une cellule et d’identifier les gènes les plus exprimés et les plus régulés[6] (Figure 2).
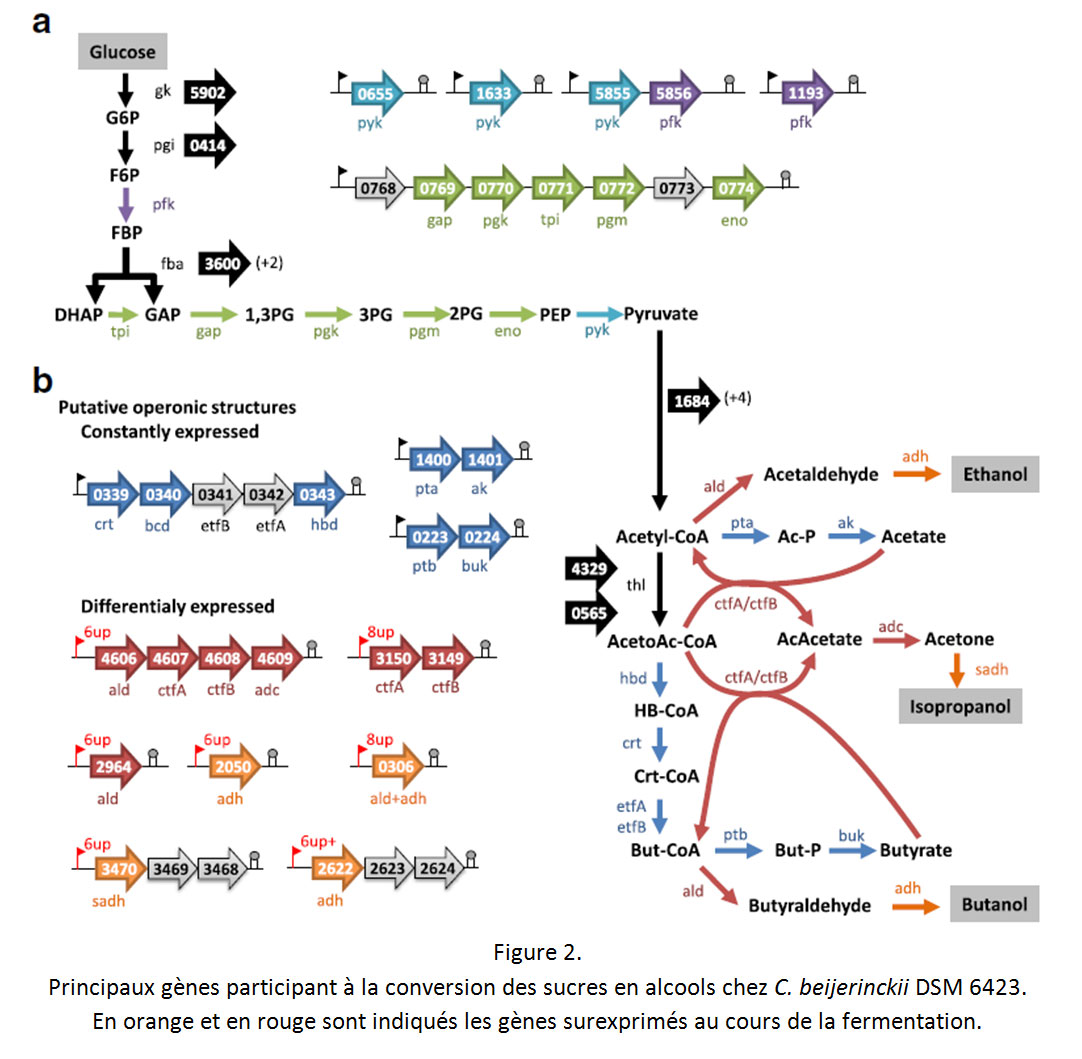
Comme attendu, on retrouve dans ce transcriptome des structures génétiques équivalentes à celles déjà décrites chez d’autres souches de Clostridium utilisées dans les procédés de type ABE. En revanche, on a pu observer que certains mécanismes de régulation de l’expression des gènes sont propres à cette souche, notamment en ce qui concerne les dernières étapes métaboliques aboutissant à la production des alcools.
Ceci nous conforte dans l’idée que le métabolisme des souches IBE se distingue de celui des souches ABE référencées dans la littérature, et nous incite fortement à poursuivre notre acquisition de données -omiques sur cette souche naturellement productrice d’isopropanol.
- Un explosif utilisé notamment par l’armée britannique lors de la première guerre mondiale.
- Élément génétique du cytoplasme (séparé et indépendant du chromosome), hébergé par un hôte bactérien et dont la réplication est autonome.
- Acide désoxyribonucléique : molécule support de l'information génétique héréditaire.
- Le transcriptome est l'ensemble des ARN issus de la transcription du génome.
- Acide ribonucléique : essentiel dans le transport du message génétique et la synthèse des protéines.
- Les gènes régulés sont ceux qui sont exprimés à certains moments et dans certaines conditions (différentiellement en fonction du type de cellule), en étant transcrits grâce à des facteurs spécifiques.
Contact scientifique : nicolas.lopes-ferreira@ifpen.fr
Référence
Máté de Gérando H., Wasels F., Bisson A., Clément B., Bidard F., Jourdier E., López-Contreras A-M., Lopes Ferreira N., Genome and transcriptome of the natural isopropanol producer Clostridium beijerinckii DSM6423 2018. BMC Genomics, 19: 242.
>> https://doi.org/10.1186/s12864-018-4636-7






